腫瘍病態学(協力)分子腫瘍学
研究室概要
当研究室は、ゲノムとRNAの作動原理の詳細を明らかにし、その精度の高い理解に基づいて、がんのメカニズムを明らかにし新たな治療法を見出すことを目指しています。遺伝子がどのように機能するのか?この基本的な問いは、遺伝子と環境の相互作用によっておきる「がん」などの多くの疾病のメカニズムを理解する上で非常に重要です。当研究室では、次世代シーケンサー・バイオインフォマティクス・ゲノム編集などの技術躍進に基づく大規模データ群の統合的解析を通じて、ゲノム・エピゲノム・転写・RNAネットワークの関係性を丹念に読み解き、遺伝子制御の作動原理や疾患のメカニズムを探索する幅広い研究を推進しています。さらに、大規模データ群の統合的解析を通じて得られた包括的生命情報を基盤として、「ゲノムの病気」であるがんに対する新たなプレシジョン医療の基本コンセプトを創出すること、そして、これを可能にする新たな方法論・技術を構築することを目標にしています。
研究プロジェクト
1. マイクロRNAとがん
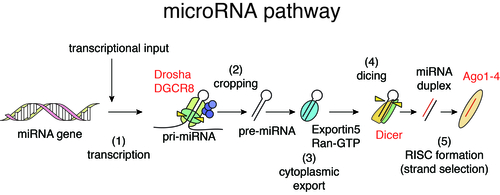
多くの遺伝子は、DNAからRNAがつくられ、RNAからタンパク質がつくられる、というセントラルドグマに沿って、機能しています。一方で、タンパク質をコードするゲノム領域以外の広範なゲノム領域で転写がおき、タンパク質へと翻訳されない多数のRNA(ノンコーディングRNA)が産生されていることが知られています。マイクロRNAは、そのようなノンコーディングRNAの代表の1つで、21~25塩基程度の低分子RNAです。マイクロRNAの存在は1993年に報告されていましたが、1998年のFire,MelloらによるRNA干渉の報告を起点として、同様の分子の検索が促され分野の研究が飛躍的に進んできました。
哺乳類細胞では、マイクロRNAは、マイクロRNAの前駆体を含む長いRNAがDrosha、DicerといったRNA切断酵素によって短く切断され、最終的に、小さなRNAがArgonaute(Ago)とよばれるタンパク質と複合体を形成することで機能します。Agoと結合したマイクロRNAは、シード配列と呼ばれる5’末端の7-8塩基に対して相補的塩基配列をもった標的mRNA(主に3’UTR領域)に結合し、遺伝子発現を主に抑制します。マイクロRNAによる標的mRNAの認識は配列特異的ですがシード配列がきわめて短いため、マイクロRNAと標的mRNAの対応は「多対多」であり、複雑な制御ネットワークを形成していると考えられています。マイクロRNAは正常の細胞の調節だけでなく、がんなどの様々な疾患に関係していることが知られています。私たちはこれまでにがんと、マイクロRNAの細胞内合成機構・遺伝子調節ネットワークの関係に注目し、がん抑制因子p53がマイクロRNAの細胞内での生合成を制御することなどを発見し、マイクロRNAの生合成のダイナミズムの一端を明らかにしてきました(Suzuki et al, Nature, 2009; Suzuki et al, Mol Cell, 2011)。
マイクロRNAは、多くの研究で、細胞増殖、細胞死、転移能といったがん細胞の特性そのものに影響を与えることが報告されてきました(細胞自律的な機能)。一方で、私たちは、マイクロRNAが悪性リンパ腫細胞の免疫形質を調節することで周囲のがん微小環境に影響を与えることを報告し、がんにおける細胞非自律的な機能の重要性を見出してきました(Matsuyama et al, Blood, 2011; Suzuki et al, Oncogene, 2015)。
2. システムRNA生物学
マイクロRNAは多くの標的mRNAを調節し、また、マイクロRNAやノンコーディングRNAが遺伝子制御に与える影響は全体的に弱いことが多く、このような複雑系をシステムとして理解するためにはゲノムワイドデータを俯瞰的に解析する新しい方法論の構築が重要です。これまでに、マイクロRNA・mRNAの発現の統合解析に基づく新規解析手法を開発し、この手法によってがんなどのより頑健なバイオマーカーをゲノムワイド発現解析から抽出できる可能性を明らかにしてきました(Suzuki et al, Nucleic Acid Res, 2013; Suzuki et al, Leukemia, 2013)。
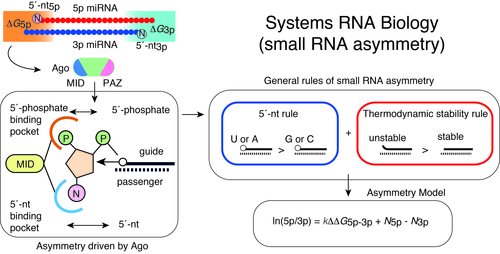
また、マイクロRNAの産生およびRNA干渉において、その起点となる二本鎖RNAからどちらのRNA鎖が機能するガイド鎖として選択されるのかは、マイクロRNAによる遺伝子制御・RNA干渉の特異性の調節において重要な問題です。この問題について、私たちは、分子生物学、生物物理学、構造生物学、バイオインフォマティクスなどを統合し、マイクロRNAの非対称性の一般的な原理、および、これに対応する分子機構を解明し、さらに、対応する数理モデルを構築することに成功してきました(Suzuki et al, Nat Struct Mol Biol, 2015; Matsuyama et al, IJMS, 2019)。特に、数理モデルの構築により、次世代シーケンサーで測定されるマイクロRNAの発現量の比を数式で予測することが可能になりました。がんでみられるマイクロRNAの変異に伴う機能の異常が、この数理モデルで説明できることも示しています。この理論的な枠組みにより、合成生物学におけるRNA回路のシミュレーションが可能となり、共同研究により合成生物学をがん免疫療法に応用可能であることを見出しています(Nissim et al, Cell, 2017)。また、このモデルはマイクロRNAの機能獲得型変異による初のヒト疾患の同定の一助ともなっています(Grigelioniene et al, Nature Medicine, 2019)。
多様な疾患で、複数のマイクロRNAの発現が異常を示すことが多く報告されています。一般的に、1つのマイクロRNAが標的遺伝子を抑制する効果は小さく、2つ以上のマイクロRNAが同じ標的遺伝子に結合することで協調的あるいは付加的に遺伝子を抑制することが重要です。最近の研究では、疾患における複数(2つ)のマイクロRNAの発現異常に注目し、統合的バイオインフォマティクスを通じて、2つのマイクロRNAが働く仕組みの多様性と、進化的変遷の特徴を明らかにしています(Kitai et al, BMC Biology, 2022)。
3. 転写の生物学(1):スーパーエンハンサーと相分離
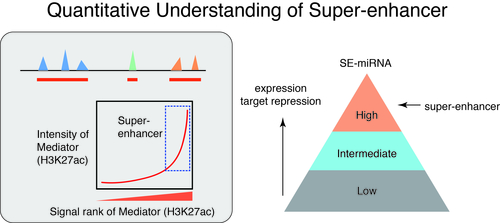
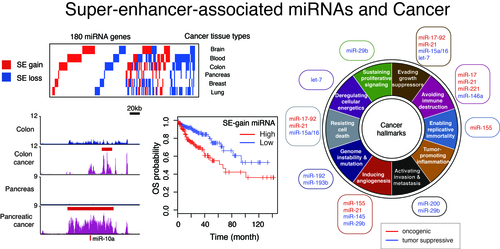
私たちの体を構成するさまざまな細胞が、同一のゲノム情報から異なる種類の細胞のアイデンティティーをどのように確立しているかは、私たちの生体を制御する遺伝子発現プログラムを理解する上で中心的な課題です。細胞種特異的なエンハンサーによる遺伝子制御は、細胞のアイデンティティーの決定において中心的な役割を果たしますが、近年、細胞種特異的な転写因子の結合領域が局所的に密集するゲノム領域であるスーパーエンハンサーが注目を集めています。私たちは、RNAに注目することで、スーパーエンハンサーの機能について研究を進めてきました。これまでに、スーパーエンハンサーとマイクロRNAの関係に注目し、細胞種特異的かつ進化的に保存されたマスターマイクロRNAによるRNA制御ネットワークが形作られる上で、スーパーエンハンサーが中心的な役割を持つことを定量的に明らかにし、スーパーエンハンサーコンセプトの妥当性を検証してきました(Suzuki et al, Cell, 2017)。また、この報告では、スーパーエンハンサーが転写だけでなく、RNAプロセシングのプラットフォームとして機能していることも見出しており、現在、エンハンサーと相分離の関係について注目して研究を行っています。
4. 転写の生物学(2):転写サイクルの理解
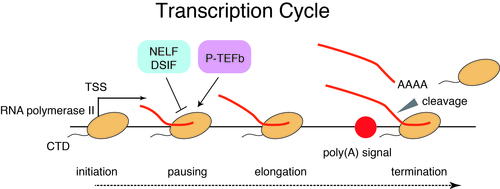
細胞のアイデンティティーを規定するエンハンサーの機能を理解するためには、エンハンサー自身でおきる転写(RNA産生)、また、エンハンサーが制御するプロモーターの転写の動態の詳細を理解することが重要です。多くのエンハンサー・プロモーターは双方向性の転写を誘導し、双方向に転写産物(RNA)を産生しています(divergent transcription)。転写を担うRNAポリメラーゼIIは、コード領域で、単純に、転写開始点からオン・オフのようにmRNAを産生しているのではなく、転写の開始・転写の一時休止(ポージング)・転写の伸長などの多段階のステップの制御を経て(転写サイクル)、最終的に機能するmRNAを産生しています。私たちは、ゲノム科学・情報科学の視点を積極的に導入することで、転写サイクルの詳細の理解を進めています。
タンパク質をコードする遺伝子はゲノム上で非常に長く分布しており、多くのプロモーターは、タンパクをコードする方向には転写が長く伸長し、逆のアンチセンス方向には早く終結する特徴を持っています。この特徴は、ゲノム上のスプライシングモチーフ (U1モチーフ) とポリアデニル化シグナル (PASモチーフ) の両方向での不均等な分布によって調節されており(U1-PAS axis)、アンチセンス側ではPASモチーフの高頻度の分布により転写の早期終結が惹起されます。私たちは、これまでに、PASモチーフにより同様の転写の早期終結がセンス側でも広範囲に起きており、この早期終結サイトとRNAポリメラーゼII (Pol II) の転写ポージングサイトがゲノム上で共局在していることを見出しています(Chiu et al, Mol Cell, 2018)。この早期終結サイト・Pol II新規転写ポージングサイトは従来知られている転写開始点直下のPol II転写ポージングサイトとは異なることを明らかにしています。
近年の大規模ながんゲノム解析の進展により、RNAスプライシング因子などの、転写やRNAプロセシングに関係する分子群の異常が新たに明らかになってきました。これらの異常の機能的意義の検証は十分にされていないことが多く、転写・RNAプロセシングの詳細の理解はがんにおける異常の理解においても重要と考えられます。急性骨髄性白血病や骨髄異形成症候群ではエンハンサーとプロモーターの相互作用に関与するコヒーシン遺伝子の変異が高頻度でみられます。最近、私たちは、共同研究により、(1)コヒーシン遺伝子の変異が転写因子の変異と協調的にゲノムの3次元構造(ループ)の異常を引き起こし骨髄異形成症候群を誘導すること、(2)コヒーシン遺伝子の変異が、広範なゲノムの3次元構造への影響とは対照的に、転写ポージングの程度が高い遺伝子群の選択的な発現低下を引き起こすことを報告しています(Ochi et al, Cancer Discovery, 2020)。
5. がんのマルチオミクス解析
大規模データ群の統合的解析に基づいて、がんの新たな分子標的を探索しています。これまでに、正常細胞・組織とがん細胞のスーパーエンハンサーの比較から,マイクロRNA遺伝子の近傍のスーパーエンハンサーに大きな変化がおき,がん細胞では,がん抑制的なマイクロRNAに関連したスーパーエンハンサーの喪失,がん促進的なマイクロRNAに関連したスーパーエンハンサーの形成がみられることを見出しています(Suzuki et al, Cell, 2017)。また、マイクロRNAの機能獲得型変異による初のヒト疾患の同定と関連して(Grigelioniene et al, Nature Medicine, 2019)、がん研究における機能ゲノミクスのビッグデータ解析の新しい方向性も報告しています(Suzuki et al, Nature Genetics, 2018)。
6. beyond
私たちは、自分たちで実施する分子生物学的方法論・アイデアに対して、臨機応変に最適なゲノムワイドデータ解析・シミュレーションを自ら構築し仮説を検証する、そして、その相互フィードバックで新しい発見をして研究を発展させてゆくという研究スタイルを構築しています。最近では、九州大学との共同研究で大規模実験データと数理シミュレーションを統合することにより、CRISPR-Cas9システムのゲノム切断活性を自在に微調整できる新技術を開発し、Cas9の過剰な活性の抑制を通じて安全性と正確な編集の効率を数百倍オーダーで高めることができる次世代型のゲノム編集プラットフォームの開発に成功しています(Kawamata et al, Nature Biomedical Engineering, 2023)。このような研究スタイルを発展させることで、新しい挑戦をしていきたいと考えています。
教員
| 構成員名 | 役職 | 所属 |
|---|---|---|
| 鈴木 洋 | 教授 | 分子腫瘍学/糖鎖生命コア研究所(iGCORE)細胞・個体制御部門(兼任) |
| 尾上 耕一 | 助教 | 分子腫瘍学 |
| 芳野 聖子 | 特任助教 | 分子腫瘍学 |
| 小松 真太郎 | 研究機関研究員 | 分子腫瘍学 |
研究実績
- 2023年
- Kawamata M, Suzuki HI, Kimura R, Suzuki A. Optimization of Cas9 activity through the addition of cytosine extensions to single-guide RNAs. Nat Biomed Eng, 2023; 7(5): 672-691.
- Suzuki HI. Roles of MicroRNAs in Disease Biology. JMAJ, 2023; 6(2): 104-113.
- 2022年
- Suzuki HI, Onimaru K. Biomolecular Condensates in Cancer Biology. Cancer Sci, 2022; 113(2): 382-391.
- Yoshino S, Suzuki HI. The molecular understanding of super-enhancer dysregulation in cancer. Nagoya J. Med. Sci, 2022; 84: 216-229.
- Miyakawa K, Miyashita N, Horie M, Terasaki Y, Tanaka H, Urushiyama H, Fukuda K, Okabe Y, Ishii T, Kuwahara N, Suzuki HI, Nagase T, Saito A. Cancer Sci, 2022; 113(11): 3932-3946.
- Shimamura Y, Furuhashi K, Tanaka A, Karasawa M, Nozaki T, Komatsu S, Watanabe K, Shimizu A, Minatoguchi S, Matsuyama M, Sawa Y, Tsuboi N, Ishimoto T, Suzuki HI, Maruyama S. Mesenchymal stem cells exert renoprotection via extracellular vesicle-mediated modulation of M2 macrophages and spleen-kidney network. Commun Biol, 2022; 5(1): 753.
- Yan M, Komatsu N, Muro R, Huynh NC, Tomofuji Y, Okada Y, Suzuki HI, Takaba H, Kitazawa R, Kitazawa S, Pluemsakunthai W, Mitsui Y, Satoh T, Okamura T, Nitta T, Im SH, Kim CJ, Kollias G, Tanaka S, Okamoto K, Tsukasaki M, Takayanagi H. ETS1 governs pathological tissue-remodeling programs in disease-associated fibroblasts. Nat Immunol, 2022; 23(9): 1330-1341.
- Kitai H, Kato N, Ogami K, Komatsu S, Watanabe Y, Yoshino S, Koshi E, Tsubota S, Funahashi Y, Maeda T, Furuhashi K, Ishimoto T, Kosugi T, Maruyama S, Kadomatsu K, Suzuki HI. Systematic characterization of seed overlap microRNA cotargeting associated with lupus pathogenesis. BMC Biol, 2022; 20(1): 248.
- 2021年
- Ogami K, Suzuki HI. Nuclear RNA Exosome and Pervasive Transcription: Dual Sculptors of Genome Function. Int J Mol Sci, 2021; 22(24): 13401.
- 2020年
- Miyashita N, Horie M, Suzuki HI, Saito M, Mikami Y, Okuda K, Boucher RC, Suzukawa M, Hebisawa A, Saito A, Nagase T. FOXL1 Regulates Lung Fibroblast Function via Multiple Mechanisms. Am J Respir Cell Mol Biol. 2020; 63(6): 831-842.
- Li Y, Que L, Fukano K, Koura M, Kitamura K, Zheng X, Kato T, Aly HH, Watashi K, Tsukuda S, Aizaki H, Watanabe N, Sato Y, Suzuki T, Suzuki HI, Hosomichi K, Kurachi M, Wakae K, Muramatsu M. MCPIP1 reduces HBV-RNA by targeting its epsilon structure. Sci Rep. 2020; 10(1): 20763.
- Tomofuji Y, Takaba H, Suzuki HI, Benlaribi R, Martinez CDP, Abe Y, Morishita Y, Okamura T, Taguchi A, Kodama T, Takayanagi H. Chd4 choreographs self-antigen expression for central immune tolerance. Nat Immunol. 2020; 21(8): 892-901.
- Ochi Y, Kon A, Sakata T, Nakagawa MM, Nakazawa N, Kakuta M, Kataoka K, Koseki H, Nakayama M, Morishita D, Tsuruyama T, Saiki R, Yoda A, Okuda R, Yoshizato T, Yoshida K, Shiozawa Y, Nannya Y, Kotani S, Kogure Y, Kakiuchi N, Nishimura T, Makishima H, Malcovati L, Yokoyama A, Takeuchi K, Sugihara E, Sato TA, Sanada M, Takaori-Kondo A, Cazzola M, Kengaku M, Miyano S, Shirahige K, Suzuki HI, Ogawa S. Combined Cohesin-RUNX1 Deficiency Synergistically Perturbs Chromatin Looping and Causes Myelodysplastic Syndromes. Cancer Discov, 2020; 10(6): 836-853.
- 2019年
- Matsuyama H, Suzuki HI. Systems and Synthetic microRNA Biology: From Biogenesis to Disease Pathogenesis. Int J Mol Sci, 2019; 21(1): 132.
- Tominaga K, Suzuki HI. TGF-β Signaling in Cellular Senescence and Aging-Related Pathology. Int J Mol Sci, 2019; 20(20): 5002.
- Kelly TJ, Suzuki HI, Zamudio JR, Suzuki M, Sharp PA. Sequestration of microRNA-mediated target repression by the Ago2-associated RNA-binding protein FAM120A. RNA, 2019; 25(10): 1291-1297.
- Akatsu Y, Takahashi N, Yoshimatsu Y, Kimuro S, Muramatsu T, Katsura A, Maishi N, Suzuki HI, Inazawa J, Hida K, Miyazono K, Watabe T. Fibroblast growth factor signals regulate transforming growth factor-β-induced endothelial-to-myofibroblast transition of tumor endothelial cells via Elk1. Mol Oncol, 2019; 13(8): 1706-1724.
- Grigelioniene G, Suzuki HI, Taylan F, Mirzamohammadi F, Borochowitz ZU, Ayturk UM, Tzur S, Horemuzova E, Lindstrand A, Weis MA, Grigelionis G, Hammarsjö A, Marsk E, Nordgren A, Nordenskjöld M, Eyre DR, Warman ML, Nishimura G, Sharp PA, Kobayashi T. Gain-of-function mutation of microRNA-140 in human skeletal dysplasia. Nat Med, 2019; 25(4): 583-590.
- 2018年
- Souma K, Shichino S, Hashimoto S, Ueha S, Tsukui T, Nakajima T, Suzuki HI, Shand FHW, Inagaki Y, Nagase T, Matsushima K. Lung fibroblasts express a miR-19a-19b-20a sub-cluster to suppress TGF-β-associated fibroblast activation in murine pulmonary fibrosis. Sci Rep, 2018; 8(1): 16642.
- Gao A, Shrinivas K, Lepeudry P, Suzuki HI, Sharp PA, Chakraborty AK. Evolution of weak cooperative interactions for biological specificity. Proc Natl Acad Sci U S A, 2018; 115(47): E11053-E11060.
- Suzuki HI, Horie M, Mihira H, Saito A. Molecular Analysis of Endothelial-mesenchymal Transition Induced by Transforming Growth Factor-β Signaling. J Vis Exp, 2018; (138): 57577.
- Miyashita N, Horie M, Suzuki HI, Yoshihara M, Djureinovic D, Persson J, Brunnström H, Lindskog C, Elfving H, Micke P, Saito A, Nagase T. An Integrative Analysis of Transcriptome and Epigenome Features of ASCL1-Positive Lung Adenocarcinomas. J Thorac Oncol, 2018; 13(11): 1676-1691.
- Suzuki HI. MicroRNA Control of TGF-β Signaling. Int J Mol Sci, 2019; 19(7): 1901.
- Suzuki HI, Spengler RM, Grigelioniene G, Kobayashi T, Sharp PA. Deconvolution of seed and RNA-binding protein crosstalk in RNAi-based functional genomics. Nat Genet, 2018; 50(5): 657-661.
- Chiu AC, Suzuki HI, Wu X, Mahat DB, Kriz AJ, Sharp PA. Transcriptional Pause Sites Delineate Stable Nucleosome-Associated Premature Polyadenylation Suppressed by U1 snRNP. Mol Cell, 2018; 69(4): 648-663.
- Lai HH, Li JN, Wang MY, Huang HY, Croce CM, Sun HL, Lyu YJ, Kang JW, Chiu CF, Hung MC, Suzuki HI, Chen PS. HIF-1α promotes autophagic proteolysis of Dicer and enhances tumor metastasis. J Clin Invest, 2018; 128(2): 625-643.
- Horie M, Miyashita N, Mikami Y, Noguchi S, Yamauchi Y, Suzukawa M, Fukami T, Ohta K, Asano Y, Sato S, Yamaguchi Y, Ohshima M, Suzuki HI, Saito A, Nagase T. TBX4 is involved in the super-enhancer-driven transcriptional programs underlying features specific to lung fibroblasts. Am J Physiol Lung Cell Mol Physiol, 2018; 314(1): L177-L191.
- 2017年
- Nissim L, Wu MR, Pery E, Binder-Nissim A, Suzuki HI, Stupp D, Wehrspaun C, Tabach Y, Sharp PA, Lu TK. Synthetic RNA-Based Immunomodulatory Gene Circuits for Cancer Immunotherapy. Cell, 2017; 171(5): 1138-1150.
- Suzuki HI, Katsura A, Mihira H, Horie M, Saito A, Miyazono K. Regulation of TGF-β-mediated endothelial-mesenchymal transition by microRNA-27. J Biochem, 2017; 161(5): 417-420.
- Suzuki HI, Young RA, Sharp PA. Super-Enhancer-Mediated RNA Processing Revealed by Integrative MicroRNA Network Analysis. Cell, 2017; 168(6): 1000-1014.
- 2016年
- Horie M, Saito A, Ohshima M, Suzuki HI, Nagase T. YAP and TAZ modulate cell phenotype in a subset of small cell lung cancer. Cancer Sci, 2016; 107(12): 1755-1766.
- Katsura A, Suzuki HI, Ueno T, Mihira H, Yamazaki T, Yasuda T, Watabe T, Mano H, Yamada Y, Miyazono K. MicroRNA-31 is a positive modulator of endothelial-mesenchymal transition and associated secretory phenotype induced by TGF-β. Genes Cells, 2016; 21(1): 99-116.
- 2015年
- Shichino S, Abe J, Ueha S, Otsuji M, Tsukui T, Kosugi-Kanaya M, Shand FH, Hashimoto S, Suzuki HI, Morikawa T, Inagaki Y, Matsushima K. Reduced supply of monocyte-derived macrophages leads to a transition from nodular to diffuse lesions and tissue cell activation in silica-induced pulmonary fibrosis in mice. Am J Pathol, 2015; 185(11): 2923-2938.
- Sugimoto K, Suzuki HI, Fujimura T, Ono A, Kaga N, Isobe Y, Sasaki M, Taka H, Miyazono K, Komatsu N. A clinically attainable dose of L-asparaginase targets glutamine addiction in lymphoid cell lines. Cancer Sci, 2015; 106(11): 1534-1543.
- Suzuki HI, Katsura A, Yasuda T, Ueno T, Mano H, Sugimoto K, Miyazono K. Small-RNA asymmetry is directly driven by mammalian Argonautes. Nat Struct Mol Biol, 2015; 22(7): 512-521.
- Suzuki HI, Katsura A, Miyazono K. A role of uridylation pathway for blockade of let-7 microRNA biogenesis by Lin28B. Cancer Sci, 2015; 106(9): 1174-1181.
- Itami S, Eguchi Y, Mizutani T, Aoki E, Ohgi T, Kuroda M, Ochiya T, Kato N, Suzuki HI, Kawada N, Murakami Y. Control of HCV Replication With iMIRs, a Novel Anti-RNAi Agent. Mol Ther Nucleic Acids, 2015; 4(1): e219.
- Suzuki HI, Katsura A, Matsuyama H, Miyazono K. MicroRNA regulons in tumor microenvironment. Oncogene, 2015; 34(24): 3085-3094.
- 2014年
- Horie M, Saito A, Noguchi S, Yamaguchi Y, Ohshima M, Morishita Y, Suzuki HI, Kohyama T, Nagase T. Differential knockdown of TGF-β ligands in a three-dimensional co-culture tumor- stromal interaction model of lung cancer. BMC Cancer, 2014; 14: 580.
- Noguchi S, Saito A, Horie M, Mikami Y, Suzuki HI, Morishita Y, Ohshima M, Abiko Y, Mattsson JS, König H, Lohr M, Edlund K, Botling J, Micke P, Nagase T. An integrative analysis of the tumorigenic role of TAZ in human non-small cell lung cancer. Clin Cancer Res, 2014; 20(17): 4660-4672.
- Tanaka M, Suzuki HI, Shibahara J, Kunita A, Isagawa T, Yoshimi A, Kurokawa M, Miyazono K, Aburatani H, Ishikawa S, Fukayama M. EVI1 oncogene promotes KRAS pathway through suppression of microRNA-96 in pancreatic carcinogenesis. Oncogene, 2014; 33(19): 2454-2463.
- 2013年
- Yoshimatsu Y, Lee YG, Akatsu Y, Taguchi L, Suzuki HI, Cunha SI, Maruyama K, Suzuki Y, Yamazaki T, Katsura A, Oh SP, Zimmers TA, Lee SJ, Pietras K, Koh GY, Miyazono K, Watabe T. Bone morphogenetic protein-9 inhibits lymphatic vessel formation via activin receptor-like kinase 1 during development and cancer progression. Proc Natl Acad Sci U S A, 2013; 110(47): 18940-18945.
- Suzuki HI, Matsuyama H, Noguchi M, Yao T, Komatsu N, Mano H, Sugimoto K, Miyazono K. Computational dissection of distinct microRNA activity signatures associated with peripheral T cell lymphoma subtypes. Leukemia, 2013; 27(10): 2107-2111.
- Saito A, Suzuki HI, Horie M, Ohshima M, Morishita Y, Abiko Y, Nagase T. An integrated expression profiling reveals target genes of TGF-β and TNF-α possibly mediated by microRNAs in lung cancer cells. PLoS One, 2013; 8(2): e56587.
- Suzuki HI, Mihira H, Watabe T, Sugimoto K, Miyazono K. Widespread inference of weighted microRNA-mediated gene regulation in cancer transcriptome analysis. Nucleic Acids Res, 2013; 41(5): e62.
- Suzuki HI, Miyazono K. p53 actions on microRNA expression and maturation pathway. Methods Mol Biol, 2013; 962: 165-181.
- Liang C, Xiong K, Szulwach KE, Zhang Y, Wang Z, Peng J, Fu M, Jin P, Suzuki HI, Liu Q.. Sjogren syndrome antigen B (SSB)/La promotes global microRNA expression by binding microRNA precursors through stem-loop recognition. J Biol Chem, 2013; 288(1): 723-736.
- 2012年
- Suzuki HI, Miyazono K. Control of MicroRNA Maturation by p53 Tumor Suppressor and MCPIP1 Ribonuclease. The Enzymes, 2012; 32: 163-183.
- Nishimori H, Ehata S, Suzuki HI, Katsuno Y, Miyazono K. Prostate cancer cells and bone stromal cells mutually interact with each other through bone morphogenetic protein-mediated signals. J Biol Chem, 2012; 287(24): 20037-20046.
- Mihira H, Suzuki HI, Akatsu Y, Yoshimatsu Y, Igarashi T, Miyazono K, Watabe T. TGF-β-induced mesenchymal transition of MS-1 endothelial cells requires Smad-dependent cooperative activation of Rho signals and MRTF-A. J Biochem, 2012; 151(2): 145-156.
- 2011年
- Suzuki HI, Arase M, Matsuyama H, Choi YL, Ueno T, Mano H, Sugimoto K, Miyazono K. MCPIP1 ribonuclease antagonizes dicer and terminates microRNA biogenesis through precursor microRNA degradation. Mol Cell, 2011; 44(3): 424-436.
- Matsuyama H, Suzuki HI, Nishimori H, Noguchi M, Yao T, Komatsu N, Mano H, Sugimoto K, Miyazono K. miR-135b mediates NPM-ALK-driven oncogenicity and renders IL-17-producing immunophenotype to anaplastic large cell lymphoma. Blood, 2011; 118(26): 6881-6892.
- Mizutani A, Koinuma D, Tsutsumi S, Kamimura N, Morikawa M, Suzuki HI, Imamura T, Miyazono K, Aburatani H. Cell type-specific target selection by combinatorial binding of Smad2/3 proteins and hepatocyte nuclear factor 4alpha in HepG2 cells. J Biol Chem, 2011; 286(34): 29848-29860.
- Suzuki HI, Miyazono K. Emerging complexity of microRNA generation cascades. J Biochem, 2011; 149(1): 15-25.
- 2010年
- Suzuki HI, Miyazono K. Dynamics of microRNA biogenesis: crosstalk between p53 network and microRNA processing pathway. J Mol Med (Berl), 2010; 88(11): 1085-1094.
- Suzuki HI, Kiyono K, Miyazono K. Regulation of autophagy by transforming growth factor-β (TGF-β) signaling. Autophagy, 2010; 6(5): 645-647.
- Hosoi M, Nannya Y, Sasaki T, Suzuki HI, Ueda K, Tsujino T, Isayama H, Takahashi T, Koike K, Kurokawa M. Biliary cast syndrome and benign biliary stricture as complications of allogeneic hematopoietic stem cell transplantation. Ann Hematol, 2010; 89(12): 1287-1289.
- Suzuki HI, Hosoya N, Miyagawa K, Ota S, Nakashima H, Makita N, Kurokawa M. Erdheim-Chester disease: multisystem involvement and management with interferon-alpha. Leuk Res, 2010; 34(1): e21-e24.
- 2009年
- Kiyono K, Suzuki HI, Matsuyama H, Morishita Y, Komuro A, Kano MR, Sugimoto K, Miyazono K. Autophagy is activated by TGF-beta and potentiates TGF-beta-mediated growth inhibition in human hepatocellular carcinoma cells. Cancer Res, 2009; 69(23): 8844-8852.
- Suzuki HI, Yamagata K, Sugimoto K, Iwamoto T, Kato S, Miyazono K. Modulation of microRNA processing by p53. Nature, 2009; 460(7254): 529-533.
- Kiyono K, Suzuki HI, Morishita Y, Komuro A, Iwata C, Yashiro M, Hirakawa K, Kano MR, Miyazono K. c-Ski overexpression promotes tumor growth and angiogenesis through inhibition of transforming growth factor-beta signaling in diffuse-type gastric carcinoma. Cancer Sci, 2009; 100(10): 1809-1816.
- Komuro A, Yashiro M, Iwata C, Morishita Y, Johansson E, Matsumoto Y, Watanabe A, Aburatani H, Miyoshi H, Kiyono K, Shirai YT, Suzuki HI, Hirakawa K, Kano MR, Miyazono K. Diffuse-type gastric carcinoma: progression, angiogenesis, and transforming growth factor beta signaling. J Natl Cancer Inst, 2009; 101(8): 592-604.
- 2008年
- Suzuki H, Tsurita G, Ishihara S, Akahane M, Kitayama J, Nagawa H. Resovist-enhanced MRI for preoperative assessment of colorectal hepatic metastases: a case of multiple bile duct hamartomas associated with colon cancer. Case Rep Gastroenterol, 2008; 2(3): 509-516.
- Suzuki HI, Izutsu K, Watanabe T, Oshima K, Kanda Y, Motokura T, Chiba S, Kurokawa M. Late-onset pneumatosis cystoides intestinalis associated with non-infectious pulmonary complications after allogeneic hematopoietic stem cell transplantation. Int J Hematol, 2008; 88(1): 116-118.
- Suzuki HI, Suzuki T, Kamijo A, Oota S, Sato H, Hangaishi A, Takahashi T, Kanda Y, Motokura T, Chiba S, Kurokawa M. Antileukemic immunity associated with antineutrophil antibody production after allogeneic hematopoietic SCT for myeloid/NK-cell precursor acute leukemia. Bone Marrow Transplant, 2008; 42(4): 285-287.
- Suzuki HI, Hangaishi A, Hosoya N, Watanabe T, Kanda Y, Motokura T, Chiba S, Kurokawa M. Herpes simplex encephalitis and subsequent cytomegalovirus encephalitis after chemoradiotherapy for central nervous system lymphoma: a case report and literature review. Int J Hematol, 2008; 87(5): 538-541.
- Suzuki HI, Asai T, Tamaki Z, Hangaishi A, Chiba S, Kurokawa M. Drug-induced hypersensitivity syndrome with rapid hematopoietic reconstitution during treatment for acute myeloid leukemia. Haematologica, 2008; 93(3): 469-470.
- Oka M, Iwata C, Suzuki HI, Kiyono K, Morishita Y, Watabe T, Komuro A, Kano MR, Miyazono K. Inhibition of endogenous TGF-beta signaling enhances lymphangiogenesis. Blood, 2008; 111(9): 4571-4579.
- Suzuki HI, Asai T, Okada K, Kazuyama Y, Takahashi T, Kanda Y, Chiba S, Kurokawa M. Disseminated adenovirus disease by multiple adenovirus serotypes following allogeneic hematopoietic stem cell transplantation. Biol Blood Marrow Transplant, 2008; 14(3): 353-355.
- 2007年
- Ogawa K, Saito A, Matsui H, Suzuki H, Ohtsuka S, Shimosato D, Morishita Y, Watabe T, Niwa H, Miyazono K. Activin-Nodal signaling is involved in propagation of mouse embryonic stem cells. J Cell Sci, 2007; 120: 55-65.
- 2002年
- Takayanagi H, Kim S, Matsuo K, Suzuki H, Suzuki T, Sato K, Yokochi T, Oda H, Nakamura K, Ida N, Wagner EF, Taniguchi T. RANKL maintains bone homeostasis through c-Fos-dependent induction of interferon-beta. Nature, 2002; 416(6882): 744-749.
研究キーワード
がん、ゲノム、RNA、マイクロRNA、ノンコーディングRNA、転写、エピゲノム、エンハンサー、スーパーエンハンサー、相分離、次世代シーケンサー、バイオインフォマティクス、ゲノム編集、遺伝子スクリーニング、シングルセル解析
大学院生
ともにサイエンスに取り組み、世界と向き合う、大学院生を募集中です。修士課程および博士課程の大学院生を受け入れています。興味のある方は鈴木まで気軽にご連絡ください。ラボの見学や進路相談など、いつでも歓迎です。
修士課程の大学院生は、CIBoG(卓越大学院プログラム)によるサポートに申請することが可能です。博士課程の大学院生は、CIBoG(卓越大学院プログラム)、名古屋大学融合フロンティアフェローシップ事業、東海国立大学機構融合フロンティア次世代研究事業などのサポートに申請することができます。また、日本学術振興会特別研究員申請のサポートも受けることができます。
2023年度
修士課程:願書受付期間
2023年7月10日(月)~7月14日(金)
博士課程:願書受付期間
第1回 2023年6月5日(月)~6月9日(金)
第2回 2023年11月13日(月)~11月17日(金)
医学部学生
当研究室での研究に興味のある方は気軽にご連絡ください。
研究員・研究スタッフ募集
当研究室での研究に興味のある方は気軽にご連絡ください(随時)。現在、助教および特任助教の公募を行なっています(2023年6月)。
連絡先
〒466-8550
名古屋市昭和区鶴舞町65 医系研究棟3号館5階
名古屋大学大学院医学系研究科分子腫瘍学
鈴木 洋
E-mail: hisuzuki@med.nagoya-u.ac.jp